Vol. 2° - IV.5.
ULTRASTRUTTURA
E ORGANIZZAZIONE MOLECOLARE
DEI CROMOSOMI
5.1. Struttura in sezioni ultrasottili
Fibre
di cromatina:
il cromosoma è costituito da fibre cromatiniche spiralizzate e accollate le
une alle altre. Dato che la sezione è ultrasottile, le fibre possono essere
seguite solo per alcuni tratti, troppo brevi per fornire un’idea della loro
disposizione.
Cinetocore:
è formato da un elemento lineare, denso, circondato da ambo i lati da una
zona fibrillare meno densa. Le estremità dei microtubuli del fuso si portano
nella zona fibrillare e penetrano fino all’elemento denso, formato da una
coppia di fibrille avvolte ad elica, con diametro variabile da 5 a 8 nm. La
regione meno densa è occupata da anse, adese con la loro estremità all’elemento
denso.
5.2. Organizzazione molecolare
Contrariamente a quanto è
accaduto per svariati organuli citoplasmatici, il microscopio elettronico non
è stato decisivo per la comprensione dei meccanismi che portano la cromatina
ad organizzarsi in cromosomi, e non ha neppure permesso un’interpretazione
soddisfacente dell’ultrastruttura cromosomica. Come per le citomembrane,
sono stati proposti modelli di organizzazione molecolare cromosomica che sono
ancora in fieri, perché sono
necessari altri dati per essere considerati modelli definitivi.
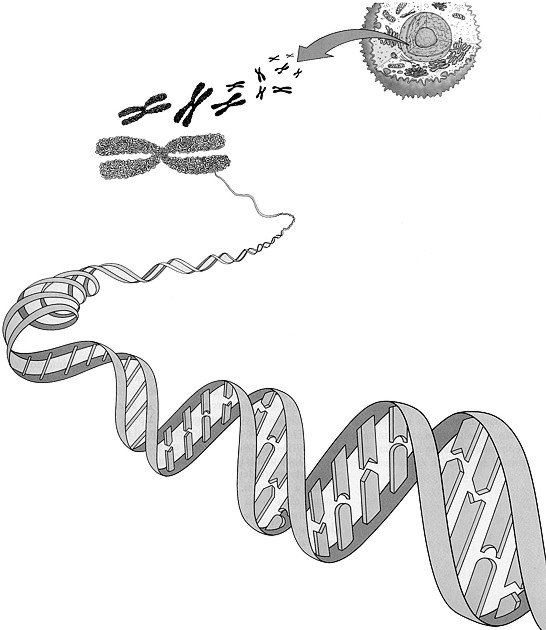
Fig. IV. 5 - Il DNA a doppia elica è contenuto nei cromosomi della cellula
Alla base dell’organizzazione molecolare dei cromosomi probabilmente si trovano le catene di nucleosomi della cromatina, i cui rapporti con le proteine non-istoniche sono complessi. Ogni catena contiene una molecola di DNA di lunghezza enorme rispetto a quella del cromosoma, valutabile in mm o cm, mentre di solito un cromosoma non è più lungo di qualche mm o decina di mm.
Esistono vari indizi per
ritenere che ogni cromosoma contenga una sola molecola lineare di DNA, che cioè esso
costituisca una struttura uninemale
[1]
.
Le prove si ricavano sia dal controllo di come avviene la duplicazione del DNA
della cromatina, sia dall’esame della struttura di particolari cromosomi
molto despiralizzati, come quelli a spazzola
delle uova di Anfibi. Sono quindi cadute le ipotesi che attribuivano ai
cromosomi una struttura polinemica,
ossia fatta da più filamenti distinti e paralleli di DNA.
Nella cellula vivente la cromatina non si trova sotto forma di nucleofilamento a collana di perle, in quanto è mantenuta in uno stato maggiormente condensato. I dettagli di questi livelli superiori di ripiegamento, osservabili soprattutto nei cromosomi, non sono ancora del tutto chiariti, come non lo sono anche i particolari relativi ai cambiamenti nel ripiegamento della cromatina che si verificano man mano che una cellula passa dall’interfase alla mitosi o alla meiosi e nuovamente all’interfase.
Esiste un accordo generale, derivato dalle osservazioni
microscopiche, che la fibra
di cromatina di 30 nm rappresenti il livello successivo di ripiegamento
al di sopra del nucleosoma. Man mano che procede l’avvolgimento della
cromatina, i singoli nucleosomi non sono più riconoscibili e quindi la loro
disposizione nella fibra di 30 nm diventa di più difficile definizione.
La
fibra di 30 nm non determina un impacchettamento della cromatina
sufficiente a spiegare il grado di condensazione dei cromosomi nel nucleo.
Vale a dire, un cromosoma umano medio, sotto forma di fibra di 30 nm, si
estenderebbe per circa 1 mm e, come tale, sarebbe 200 volte più lungo del
diametro del nucleo, pari a circa 5 mm.
Deve quindi verificarsi un ulteriore ripiegamento della fibra di cromatina.
5.3. Modello a fibrilla elicoidale
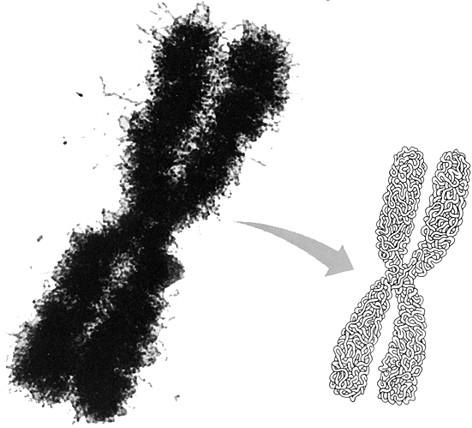
Fig.
IV. 6 - Fotografia al microscopio elettronico
di un
cromosoma metafasico ingrandito 30.000 volte
A destra è
rappresentata
la struttura a fibrilla elicoidale secondo l’ipotesi di Du Praw.
Il diametro di un cromosoma, ben visibile anche al microscopio luce, è di qualche mm; la fibrilla composta da DNA e istoni, che nell’eterocromatina è larga 25 nm (ossia 1/40 di mm), è dunque ripiegata varie volte su se stessa anche in senso trasversale al cromosoma.
Seguendo l’ipotesi di Du Praw (1965) si ammette che, all’interno di un cromosoma, la
fibrilla di DNA-proteine, la
fibrilla elementare, formi una
matassa più o meno regolare, ripiegandosi varie volte in lunghezza e
larghezza, fino a costituire un grossolano filamento di notevoli
dimensioni, il cromonema, che,
ripiegato ad elica, forma l’asse del cromosoma.
Questo modello detto a
fibrilla elicoidale si basa su osservazioni in microscopia elettronica, in
base alle quali si può dimostrare che un cromosoma risulta composto da una
fibrilla, intrecciata su se stessa in maniera complessa, con anse sia
trasversali che longitudinali, la quale ha un diametro minimo di 25 nm. Questa
fibrilla forma pacchetti di diametro
sempre maggiore (50, 200, 600 nm) fino a costituire le minor e major coils
visibili anche al microscopio luce lungo l’asse cromosomico.
5.4. Modello dei dominî ad ansa
Recentemente è stato proposto
un modello di struttura del cromosoma che tiene conto della localizzazione,
oltre che del DNA e degli istoni, anche di una quota delle proteine
non-istoniche della cromatina. La novità di questo modello consiste nell’aver
messo in evidenza che il cromosoma non è costituito soltanto da un filamento
ripiegato di DNA e proteine, ma che possiede uno scheletro o impalcatura centrale
o scaffold, di natura proteica,
relativamente indipendente dal filamento di DNA e istoni, detto filamento
nucleosomiale.
Tale modello, a loops
radiali (loops = anse)
o a microanse,
si basa sull’osservazione, in microscopia elettronica, di cromosomi privati
degli istoni e di circa il 70% delle proteine non-istoniche per mezzo di
blandi detergenti o di soluzioni saline particolari. Così trattati, i
cromosomi si presentano come strutture che conservano la forma visibile anche
al microscopio luce, ma il cui DNA si spande lateralmente in anse della
lunghezza media di 10-30 mm (fino a 60 mm). La morfologia cromosomica
viene mantenuta dalla presenza di un’impalcatura proteica di natura
fibrillare, che forma una rete centrale dalla quale si dipartono le anse
laterali di DNA.
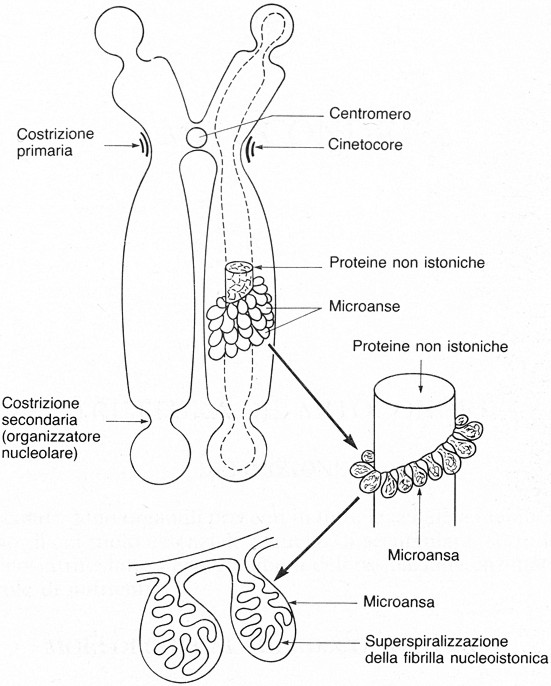
Fig.
IV. 7 - Struttura di un cromosoma metafasico
secondo il modello dei domini ad
ansa o a loops radiali
L’impalcatura, o scaffold,
è costituita da una trentina di proteine non-istoniche in gran parte comuni
alla matrice nucleare. Il DNA delle anse risulta espanso lateralmente, perché
col trattamento sono stati estratti i core
istonici nucleosomiali, che normalmente tengono impacchettato il DNA in
strutture che sporgono solo di 3-4 mm
dall’asse cromosomico.
Le anse di DNA iniziano e terminano in punti dell’impalcatura
proteica contigui fra loro. Le varie anse sono unite a tale impalcatura da
brevi tratti longitudinali di DNA: il DNA di un cromosoma è quindi un
filamento continuo, in piccola parte aderente allo scaffold
proteico, in misura maggiore espanso lateralmente in loops.
Probabilmente questo modello a loops radiali non è definitivo, perché resta da chiarire la
posizione e il ruolo di quella larga frazione di proteine non-istoniche
allontanata dal trattamento. Esso però ha vari vantaggi rispetto al modello a
fibra ripiegata proposto da Du Praw:
o
si accorda bene con le osservazioni sulla struttura dei
cromosomi a spazzola ovocitari, i
quali hanno un’organizzazione a loops
laterali simile a quella ora descritta
o
si accorda pure con le osservazioni sulla struttura dei
genofori procariotici, che dimostrano come il DNA circolare dei batteri formi loops
radiali attorno a uno stroma centrale fatto da RNA e proteine
o
i loops radiali
di DNA hanno una lunghezza che corrisponde bene a quella delle unità di
replicazione o repliconi,
individuabili, con esperienze di marcatura, sui cromosomi eucariotici: ogni
loop
sarebbe quindi un
replicone
o
lo
scaffold centrale al cromosoma,
fatto di proteine strutturali, trova un riscontro, a livello dei cromosomi
meiotici, in una struttura nota da tempo col nome di complesso
sinaptinemale: ciò dimostra che strutture scheletriche si rinvengono
comunque nei cromosomi eucariotici e costituiscono probabilmente il risultato
della riaggregazione della rete fibrillare endonucleare che è visibile solo
nel nucleo intercinetico.
Secondo alcune ipotesi i loops radiali conterrebbero le frazioni trascrivibili del genoma, mentre i tratti di DNA aderenti allo scaffold, con funzione eminentemente strutturale, accoglierebbero le frazioni altamente ripetitive, e il loro ruolo strutturale risulterebbe meglio spiegato in base al modello dei dominî ad ansa.