Vol. 2° - XIII.8.
ASSOCIAZIONE TRA GENI
Fig.
XIII. 2 - Mutazioni oculari in Drosophila melanogaster. In
ambedue i soggetti gli occhi sono più piccoli della norma e le ali presentano
venatura diversa. Questo moscerino, che abbiamo spesso scacciato dalla frutta
matura, è stato di importanza fondamentale per gli studi di genetica. E
questo moscerino è tornato alla ribalta grazie a McCall & Steller (1997)
per gli importantissimi studi sull'apoptosi cellulare.![]()
8.1. Concatenazione o Linkage
Le ghiandole salivari della Drosophila
melanogaster
[1]
,
il comune moscerino della frutta, sono provviste di cromosomi giganti sui
quali si possono agevolmente distinguere le bande evidenziate dai classici
procedimenti di citogenetica. Dobbiamo molto alla Drosofila in quanto, a
partire dal 1910, divenne il materiale favorito del biologo americano Morgan e dei suoi Allievi. Presenta, rispetto all’uomo
e ai piselli, il vantaggio di produrre una nuova generazione ogni 20 giorni,
ha solo 4 coppie di cromosomi nettamente differenziati, infine è
particolarmente soggetta alle mutazioni, per cui la scuola di Morgan ha potuto
chiarire molti interrogativi sul comportamento cromosomico.
Una prima scoperta fu il riconoscimento di una solidarietà
fra certi geni, il che spiega perché alcuni
caratteri si manifestino sempre associati: è il fenomeno del linkage
[2]
,
dovuto al fatto che questi geni sono solidali poiché sono situati sullo
stesso cromosoma. Un esempio tipico e lampante
di linkage si osserva per i caratteri legati al sesso.
8.2. I gruppi di associazione
Non sempre è possibile
assegnare i geni a un particolare cromosoma, come accade invece facilmente per
i geni portati dai cromosomi sessuali (Z e W nel pollo). Al contrario, i geni
associati, o concatenati, o linked,
appartenenti agli autosomi, spesso vengono attribuiti a specifici gruppi di associazione,
definibili in base alla caratteristica posseduta da alcuni geni di essere
ereditati non del tutto a caso né in modo tra loro indipendente, bensì a
gruppi e secondo la loro combinazione originaria. Il numero dei gruppi di
associazione di un organismo, se sono stati stabiliti con certezza, e con
altrettanta certezza sono stati mappati i geni indiziati, corrisponde al
numero aploide del corredo cromosomico.
Per il Pollo
Domestico il numero dei gruppi di associazione finora studiati con
sufficiente sicurezza non corrisponde al corredo aploide dato dalla somma dei
macrocromosomi e dei microcromosomi. Infatti i gruppi di associazione
descritti da Bitgood & Somes
[3]
,
validi fino a poco tempo fa, sono i seguenti:
q
cromosoma
sessuale W (ex gruppo VI)
q
cromosoma
sessuale Z (ex gruppo V)
q
cromosoma
1 - gruppo III
q
cromosoma
2 - forse contenente i gruppi di associazione I - II - IV
q
cromosoma
6 - gruppo VII
q
cromosoma
7 - gruppo VIII
q
gruppo
IX - discontinuo, non si tratta di un classico gruppo di linkage, ma utile per
enumerare i loci trovati sui microcromosomi
q
microcromosoma
17 - gruppo X - contiene la regione organizzatrice del nucleolo (NOR) e il
locus B per il Complesso Maggiore di Istocompatibilità (MHC).
Nell’aprile 1994, grazie alle
sempre più sofisticate metodiche di studio e agli incroci con successivi
incroci a ritroso eseguiti da Crittenden a East Lansing (USA), i gruppi di
associazione sono
saliti a 33: E1-E38, EW, EZ. I soggetti impiegati in questo studio furono un
Gallo Rosso della giungla e femmine Livorno bianca.
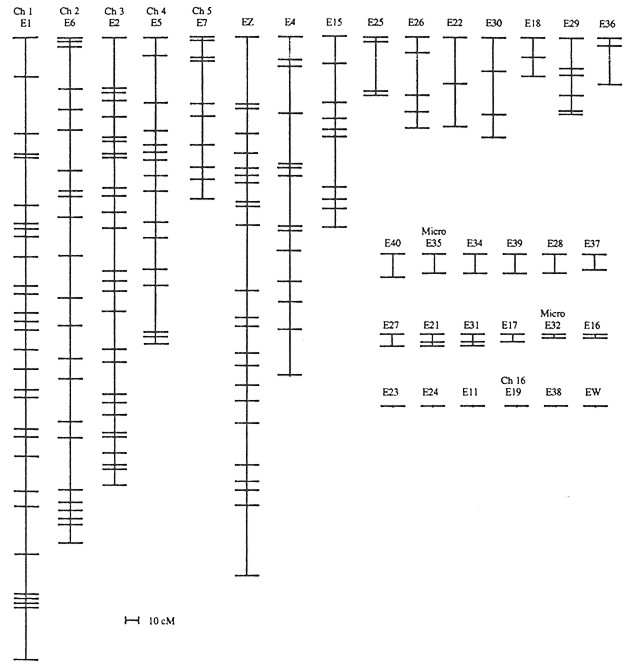
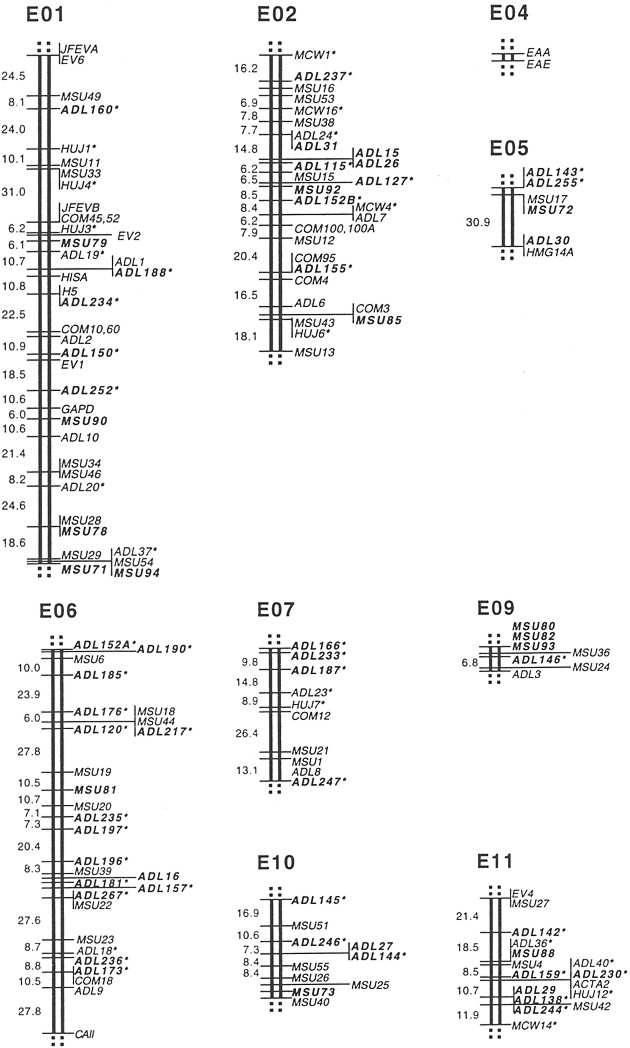
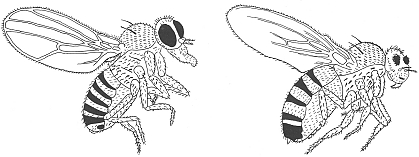
Fig. XIII. 3 – Mappa dei gruppi di associazione del
genoma del pollo. La mappa si basa su 33 gruppi di linkage - etichettati E1-E38, EW, EZ
- ed è frutto degli incroci a ritroso eseguiti a East Lansing da Crittenden
(1994). Ch1, Ch2, Ch3, Ch4, Ch5 e Ch16 sono le assegnazioni cromosomiche dei
gruppi di linkage. L’assegnazione del gruppo di linkage E2 a Ch3 è un
tentativo e si basa sulla mappazione genica comparativa.
Analizzeremo in dettaglio i gruppi di associazione in un
capitolo apposito. L’analisi del linkage
serve a determinare sperimentalmente sino a che punto i geni tendono a
rimanere uniti mentre si ricombinano. Ciò avviene con una frequenza
inversamente proporzionale alla distanza che separa tali geni fra loro.
Infatti, quanto maggiore è il grado di segregazione casuale di due geni,
tanto minore è il linkage, e
proporzionalmente maggiore è la probabilità che essi possano ricombinarsi. Quando la probabilità di ricombinazione tra due geni è pari al
50%,
si dice che la segregazione è casuale e che i geni sono indipendenti, sebbene
talvolta possano essere associati.
Mappare i geni, cioè determinarne la localizzazione, è un momento fondamentale nella comprensione del concetto di gene. La mappatura genica fornisce informazioni utili in quanto, per esempio, può indicare se i geni che lavorano in modo coordinato nel produrre un determinato fenotipo siano localizzati sullo stesso cromosoma.
L’edizione
più recente della mappa del linkage riporta un numero pari a
circa 460 loci