Lessico
Genetica di popolazioni
Il termine deriva dal greco haplóos che in parole composte dotte o scientifiche significa ‘singolo’ o ‘semplice’. Combinazione di varianti alleliche lungo un cromosoma o segmento cromosomico contenente loci in linkage disequilibrium, cioè strettamente associati tra loro. L'associazione statistica tra loci si manifesta in assenza di ricombinazione tra i loci stessi. Per quanto riguarda gli autosomi (cromosomi non sessuali) e le regioni pseudoautosomiche dei cromosomi sessuali questo può essere dovuto alla vicinanza fisica tra i loci considerati e all'assenza di hot-spot di ricombinazione tra loro.
Invece gli alleli della regione non ricombinante del cromosoma Y (NRY) sono sempre associati a formare aplotipi, così come gli alleli del genoma mitocondriale (mtDNA). Infatti queste due porzioni del genoma non ricombinano, essendo ereditate con modalità uniparentali, paterna la prima, materna la seconda.
Aplotipi differenti sono generati da un aplotipo ancestrale per effetto della mutazione ai singoli loci. I prodotti di questo meccanismo evolutivo possono essere correlati attraverso la filogenesi fino a desumere la forma ancestrale dell'aplotipo. Spesso, quando la risoluzione molecolare di un aplotipo è molto elevata, può essere utile raggruppare filogeneticamente aplotipi diversi sulla base di un comune progenitore definendo così un aplogruppo.
Applicazioni della tipizzazione aplotipica
L'analisi degli aplotipi in linkage disequilibrium costituisce uno strumento importante per il mappaggio genetico ad alta risoluzione. Gruppi di aplotipi tra loro affini evolutivamente costituiscono aplogruppi. Gli aplogruppi della regione non ricombinante del cromosoma Y (NRY) e quelli del DNA mitocondriale (mtDNA) sono particolarmente significativi in studi di filogenesi, tassonomia ed evoluzione.
La conoscenza degli aplotipi HLA è coinvolta nel rigetto dei trapianti e nella valutazione del rischio genetico di suscettibilità ad alcune patologie autoimmuni (diabete, celiachia).
Polimorfismo
a singolo nucleotide
Un polimorfismo a singolo nucleotide (spesso definito in inglese Single Nucleotide Polymorphism o SNP, pronunciato snip) è un polimorfismo (cioè una variazione a livello di una sequenza di acidi nucleici) che si presenta tra individui della stessa specie, caratterizzata da una differenza a carico di un unico nucleotide.
Ad esempio, se le sequenze individuate in due pazienti sono AAGCCTA e AAGCTTA, è presente un SNP che differenzia i due alleli C e T.
Frequenza degli SNPs
All'interno di una popolazione è possibile determinare una minor frequenza allelica, il rapporto tra la frequenza della variante più rara e quella più comune di un determinato SNP. Solitamente si guarda con maggiore attenzione a SNPs aventi minor frequenza allelica minore all'1%, trascurando nelle analisi la maggior parte degli SNPs che, anche per il loro elevatissimo numero, risultano poco maneggevoli. È importante notare che possono esistere variazioni notevoli tra popolazioni umane. Uno SNP molto comune in determinato gruppo etnico può dunque essere molto raro in un'altra popolazione.
Gli SNPs possono presentarsi all'interno di una sequenza codificante di un gene, all'interno di una regione intronica o in una regione intergenica. Gli SNPs all'interno di un gene, in ogni caso, non necessariamente modificano la sequenza aminoacidica codificata, dal momento che il codice genetico è degenerato. Uno SNP che genera in tutte le sue forme lo stesso peptide è detto sinonimo (synonymous); in caso contrario è detto non-sinonimo (non-synonymous). Gli SNPs che non si trovano in una sequenza codificante possono, in ogni caso, presentare sequenze negative sullo splicing o sul legame dei fattori di trascrizione.
Gli SNPs costituiscono il 90% di tutte le variazioni genetiche umane. SNPs con minor frequenza allelica pari a circa l'1% sono presenti ogni circa 100-300 paia di basi lungo l'intero genoma. In media, due SNPs su tre vedono una variazione tra citosina e timina.
Potenzialità degli SNPs
Lo studio degli SNPs è molto utile poiché variazioni anche di singoli nucleotidi possono influenzare lo sviluppo delle patologie o la risposta ai patogeni, agli agenti chimici, ai farmaci. Per tale motivo gli SNPs possono avere una grande importanza nello sviluppo di nuovi farmaci e nella diagnostica, in quanto consentono di conoscere l'effetto che può avere un farmaco su un individuo ancor prima della somministrazione, attraverso uno screening degli SNPs presenti nel gene responsabile della metabolizzazione del farmaco stesso; queste sono le basi della farmacogenomica. Dal momento che gli SNPs sono perlopiù ereditati di generazione in generazione, essi vengono utilizzati in alcuni studi genetici.
Individuazione degli SNPs
Un metodo utile per individuare gli SNPs è la valutazione dei cosiddetti restriction fragment length polymorphisms (polimorfismi di lunghezza dei frammenti di restrizione, o RFLP). Se un allele contiene un sito di riconoscimento per un enzima di restrizione e un altro no, la digestione dei due alleli genererà due frammenti di dimensione differente. In realtà oggi gli SNPs sono studiati principalmente attraverso i microarrays, che permettono l'analisi simultanea di centinaia di diversi SNPs e una veloce analisi elaborata da un computer.
Una comparazione del DNA mitocondriale di appartenenti alla razza umana di diverse etnie e regioni, suggerisce che tutte queste sequenze di DNA si siano evolute molecolarmente dalla sequenza di un antenato comune. In base all'assunto che un individuo erediti i mitocondri solo dalla propria madre, questa scoperta implica che tutti gli esseri umani abbiano una linea di discendenza femminile che deriva da una donna che i ricercatori hanno soprannominato Eva mitocondriale. Basandosi sulla tecnica dell'orologio molecolare che mette in correlazione il passare del tempo con la deriva genetica osservata, si ritiene che Eva sia vissuta circa 150.000 anni fa. La filogenia suggerisce che sia vissuta in Africa.
L'importanza di Eva
Anche se prende il nome dall'Eva biblica, l'Eva mitocondriale non era l'unica femmina umana del suo tempo. Fino a 20.000 individui della specie di Eva potrebbero aver vissuto nel suo tempo. Ma solo Eva avrebbe prodotto una linea ininterrotta di figlie che persiste ancor oggi. Come risultato, solo i mitocondri di Eva avrebbero discendenti nelle cellule degli esseri umani viventi. Eva sarebbe l'unica femmina della sua generazione dalla quale tutte le persone viventi discendono attraverso le loro linee materne.
L'Eva mitocondriale sarebbe il più recente antenato mitocondriale di tutti gli esseri umani viventi, ma, naturalmente, sua madre, sua nonna, e così via, erano anch'esse parte di questa linea materna.
L'Eva mitocondriale degli esseri umani attualmente viventi non era probabilmente la stessa Eva mitocondriale degli esseri umani che vissero migliaia di anni fa, o che vivranno tra qualche migliaio di anni.
Si noti che Eva non deve essere necessariamente il nostro più recente antenato comune. Comunque, poiché la riproduzione sessuata mischia il DNA dei cromosomi, la datazione di un antenato comune più recente rimane impossibile da esplorare con i mezzi attuali.
La catena degli eventi
Il fatto sorprendente che nessun'altra linea esclusivamente femminile sia sopravvissuta dal periodo di Eva, viene assunto come un effetto del caso, piuttosto che della selezione naturale. Una donna ottiene il titolo di Eva retroattivamente, attraverso una serie eccezionale di figlie. In ognuna delle sue generazioni discendenti, c'è una figlia che dà alla luce un'altra figlia. Solo quando le serie di tutte le altre contendenti vengono spezzate, Eva prende possesso del suo titolo. Come conseguenza, il premio non è assolutamente certo, perché non tutti gli individui sono testati per la discendenza e tra i miliardi di individui sulla terra, potrebbero essercene alcuni discendenti da altre linee materne.
Essenzialmente, il processo ipotizzato per cui tutte le linee di discendenza eccetto una scompaiono, è lo stesso della deriva genetica degli alleli. Come è vero per la "fissazione" o la soppiantazione di tutti gli altri alleli in base alla deriva genetica, il processo di fissazione matrilineare è molto più lento e molto più improbabile che raggiunga il completamento in un'ampia popolazione rispetto a una popolazione ridotta. Se Eva è vissuta assieme a un milione o un miliardo di altre femmine, è molto improbabile che gli antenati matrilineari di tutti gli esseri umani viventi oggi convergano su Eva (o su qualsiasi sua contemporanea).
Perché la comunità delle pari di Eva sarebbe stata così piccola? Una possibilità è che la popolazione mondiale degli esseri umani nell'epoca di Eva sia passata attraverso un collo di bottiglia. Un'altra è che Eva visse in una sottopopolazione di umani che soppiantò tutti gli altri. Una versione ancor più estrema di quest'ultimo scenario è che Eva visse poco dopo un qualsiasi evento isolante causato dalla speciazione degli esseri umani moderni (Homo sapiens). Dei resti di Homo sapiens scoperti finora, infatti, il più antico le cui ossa corrispondono a quelle degli esseri umani viventi, risale all'incirca al periodo in cui sarebbe vissuta Eva.
Relazione con Adamo
D'altra parte, ci sarebbe stato un uomo più recente, l'Adamo Y-cromosomale, che avrebbe generato una linea di maschi da cui discendono tutti gli uomini della Terra. L'Adamo Y-cromosomale sembrerebbe essere vissuto circa 75.000 anni fa, la metà dell'Eva mitocondirale. Questo significa che un altro collo di bottiglia, oltre a quello che circonda Eva, influì sulla linea di discendenza umana dopo di lei. Il fatto che il collo di bottiglia del tempo di Adamo non sembra abbia prodotto anche un antenato matrilineare di tutti gli esseri umani viventi — in altre parole, un'altra Eva — illustra che le diramazioni e scomparse nelle linee di discendenza dipendono dal caso (in alternativa, le linee di discendenza maschile potrebbero svanire più rapidamente, forse a causa di una storia di poligamia, che avrebbe permesso solo a una porzione dei maschi di produrre una discendenza). Alcuni ricercatori sostengono che prove di questo secondo collo di bottiglia esistono anche nei dati del DNA mitocondriale. È anche possibile che i dati discordanti di Eva e Adamo possano illustrare imperfezioni nella tecnica dell'orologio molecolare, che continua a subire miglioramenti.
Sfide alla teoria
Una recente sfida alla teorie dell'Eva mitocondriale è data dall'osservazione che i mitocondri dello spermatozoo vengono talvolta passati alla progenie. Altre prove suggeriscono che il DNA mitocondriale di spermatozoi e ovuli potrebbe ricombinarsi, o scambiarsi pezzi di sequenza. Quindi i mitocondri potrebbero non essere un indicatore di matrilinearità così puro come si supponeva quando venne proposta la teoria. In base a quanto frequentemente avvengono l'eredità paterna e la ricombinazione, oltre a quando avvengono, potrebbe darsi che non sia mai esistita una Eva mitocondriale. Ma gli scienziati ancora discordano sul fatto che si verifichino questi processi, e se avvengono, se lo facciano con frequenza sufficiente a escludere la possibilità di una Eva.
Eva e la teoria del fuori dall'Africa
L'Eva mitocondriale viene talvolta indicata come Eva africana, un antenato che è stato ipotizzato in base a prove fossili e del DNA. Secondo la più comune interpretazione dei dati del DNA mitocondriale, i titoli appartengono alla stessa ipotetica donna. Gli alberi genealogici (o filogenie) costruiti sulle basi del confronto del DNA mitocondriale mostrano che gli esseri umani viventi le cui linee di discendenza mitocondriale si sono ramificate per prime sono quelle degli indigeni africani, mentre le linee di discendenza delle popolazioni indigene di altri continenti si ramificano tutte dalla linea africana. I ricercatori ne concludono quindi che tutti gli esseri umani viventi discendono dagli africani, alcuni dei quali migrarono fuori dall'Africa per popolare il resto del mondo. Se l'analisi mitocondriale è corretta, allora poiché l'Eva mitocondriale rappresenta la radice dell'albero genealogico mitocondriale, deve essere vissuta prima dell'esodo e aver vissuto in Africa. Per questo molti ricercatori prendono le prove mitocondriali a supporto dell'ipotesi della singola origine o modello fuori dall'Africa.
La costruzione di alberi genealogici dai dati del DNA è comunque una scienza inesatta. I critici del modello della "genesi africana" sostengono che le prove mitocondriali possono essere spiegare altrettanto bene, se non meglio, con alberi che associano più strettamente Eva alle popolazioni indigene dell'Asia. Al 2003, comunque, a seguito di miglioramenti nella potenza di calcolo e nei metodi di determinazione degli alberi, queste critiche sono diminuite. In ogni caso, il forte supporto offerto dal DNA mitocondriale per l'ipotesi dell'origine africana potrebbe non dipendere dagli alberi. Una scoperta non soggetta a interpretazione è che la più grande diversità delle sequenze di DNA mitocondriale esiste tra gli africani. Questa diversità non si sarebbe accumulata, sostengono i ricercatori, se gli esseri umani non avessero vissuto in Africa per più tempo che in qualsiasi altra parte. Le analisi delle sequenze del cromosoma Y hanno corroborato le prove fornite dal DNA mitocondriale per l'origine africana degli ominidi.
I più recenti antenati comuni
Altri antenati comuni più recenti hanno probabilmente contribuito a diversi geni che risiedono nel DNA nucleare lineare. Ad esempio, alcuni antenati comuni più recenti potrebbero aver contribuito con un gene che specifica una delle sottounità dell'RNA nei ribosomi. Comunque, poiché la riproduzione sessuata mischia il DNA nucleare dei cromosomi forniti dai due genitori, un antenato comune più recente di Eva resta difficile da identificare con i mezzi attuali.
Nella finzione
Il romanzo e film horror giapponese Parasite Eve usa la teoria dell'Eva mitocondriale come base per una storia in cui uno scienziato fa risorgere sua moglie tramite la rigenerazione delle cellule del suo fegato, con effetti disastrosi.Il racconto Eva mitocondriale di Greg Egan tratta anch'esso di questo tema.
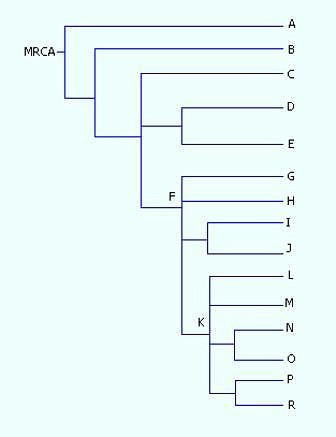
Principali
aplogruppi del cromosoma Y (lettere A-R)
correlati filogeneticamente in un albero. MRCA = Most Recent Common Ancestor
In Genetica umana, gli aplogruppi del cromosoma Y sono raggruppamenti di combinazioni di marcatori (aplotipi) definiti dalle differenze nella regione non-ricombinante del DNA del cromosoma Y (chiamato NRY da Non-Recombining Y-chromosome). Queste differenze fanno riferimento a polimorfismi biallelici (SNPs, Single Nucleotide Polymorphisms).
Il YCC (Y Chromosome Consortium) ha stabilito un sistema per definire gli aplogruppi del cromosoma Y basato sulle lettere da A a R, con ulteriori divisioni usando numeri e lettere in pedice.
Il cromosoma Y ancestrale (scherzosamente definito dagli studiosi "di Adamo") è quello appartenuto a un maschio teorico che rappresenta il più recente progenitore comune (MRCA Most Recent Common Ancestor) di tutti i maschi attuali lungo la linea patrilineare, visto che il cromosoma Y è unicamente trasmesso dal padre ai figli maschi. La stima di quando questo individuo teorico sia vissuto varia a seconda degli studi.
Aplogruppi A e B
In nessuna parte del mondo sono stati trovati lignaggi del cromosoma Y più antichi di 200.000 anni. Gli aplogruppi A e B sono considerati i più antichi e si trovano soltanto nell'Africa Sub-Sahariana o in popolazioni con la stessa origine, come per esempio gli Afroamericani portati oltreoceano con la tratta degli schiavi. Le frequenze più importanti di A si trovano tra i Boscimani, i Khung e i Sudanesi. Le frequenze più elevate di B tra i Pigmei Biaka e Mbuti. Come si evince dall'albero filogenetico degli aplogruppi del cromosoma Y, tutta la diversità moderna si è generata in Africa, per cui le popolazioni contemporanee sono discendenti degli Africani che restarono in Africa o di quelle popolazioni che emigrarono fuori dall'Africa per popolare il resto dei continenti.
La prima biforcazione dal tipo ancestrale teorico (di Adamo) deriva dalla mutazione M91 che definisce l'aplogruppo A (dal quale derivano A1, A2 e A3). Tutti gli altri aplogruppi derivano da BR (noto anche come YxA, che significa: tutti i cromosomi Y che non appartengono all'aplogruppo A). Aplogruppi C-R
Le mutazioni M168 e M294, assenti in A e B, definiscono tutti gli aplogruppi da C a R. Queste mutazioni precedettero la migrazione fuori dall'Africa, essendo presenti sia in Africa che al di fuori. Le mutazioni che caratterizzano DE (M145, M203) si verificarono in Africa orientale più di 60.000 anni fa e parteciparono alle grandi migrazioni precoci, cosicché oggi si ritrovano solo tra Esquimesi e Amerindi. L'aplogruppo E rimase originariamente in Africa e le sue più alte frequenze si riscontrano in Africa Sub-Sahariana occidentale (81%) e Etiopia (68%). Il sub-clade E1a è di origine africana e si disperse per tutto il Mediterraneo raggiungendo la frequenza del 27% in Grecia. Anche E3b si trova nel sud-Europa. L'aplogruppo D si trova soltanto in Asia, soprattutto nell'Himalaya e in Giappone, dove fu introdotto dai primi colonizzatori.
Aplogruppi G-R (sub-clade di F)
Gli aplogruppi che discendono da F rappresentano il 90% della popolazione mondiale, ma si distribuiscono quasi esclusivamente fuori dall'Africa sub-sahariana. I-J corrisponde probabilmente a un'ondata migratoria dal Medio-Oriente o all'Asia occidentale a partire da 45.000 anni fa, che si è poi diffusa in Europa con l'uomo di Cro-Magnon. L'aplogruppo G, originatosi anch'esso in Medio-Oriente, o nel Caucaso, o forse più a Est in Pakistan, intorno a 30.000 anni fa, secondo alcuni studi potrebbe essersi diffuso in Europa nel Neolitico, oppure, vista la sua forte discontinuità, aver raggiunto l'Europa già nel Paleolitico. L'aplogruppo H si originò forse in India 30-40.000 anni fa, dove persistette fino a ridiffondere in epoche storiche con i Gitani. L'aplogruppo K si originò probabilmente nell'Asia sud-occidentale e da lì si diffuse in Africa, Eurasia, Australia e Sud-Pacifico.
Aplogruppo K (L-R)
L'aplogruppo L si trova principalmente nel sud dell'Asia. L'aplogruppo M è prevalente nelle isole Papua e Nuova Guinea. Gli aplogruppi N e O comparvero 35-40.000 anni fa in Asia centrale. L'aplogruppo N si originò probabilmente in Mongolia e si diffuse fino all'estremo Oriente e la Siberia come a ovest, essendo il gruppo più comune tra i popoli uralici. L'aplogruppo O si trova in estremo Oriente e Oceania. L'aplogruppo P si trova soprattutto come sottotipi Q o R, raramente non differenziato. Si evolse probabilmente nell'Asia centrale o nella regione di Altai. Anche l'aplogruppo Q si originò nell'Asia centrale, migrò verso est e raggiunse l'America del nord attraverso lo stretto di Bering.
Aplogruppo I
L'aplogruppo I rappresenta circa un quinto dei cromosomi Y europei. È quasi esclusivo dell'Europa e pertanto si ritiene che si sia originato in quest'area prima dell'ultima glaciazione. È probabile che sia stato confinato nel rifugio balcanico durante la glaciazione e che poi si sia ridiffuso verso nord con il ritiro dei ghiacciai. Nonostante sia relativamente frequente negli Scandinavi, nei Sardi e nelle popolazioni balcaniche, questi popoli presentano subcladi differenti dell'aplogruppo I. Questo suggerisce che ognuna delle popolazioni ancestrali è oggi dominata da un particolare subclade che ha marcato una indipendente espansione della popolazione lungo diversi percorsi migratori durante e immediatamente dopo la glaciazione.
I principali subcladi dell'aplogruppo I sono:
I1a (M253, M307, P30, P40) con le più alte frequenze in Scandinavia, Islanda, e Europa nord- orientale. Nelle Isole britanniche la mutazione I1a-M253 è spesso usata come marcatore delle invasioni vichinghe o anglosassoni.
I1b (S31) che include I1b1 (P37.2), la forma più comune nei Balcani e in Sardegna (dove rappresenta l'aplogruppo più cospicuo nella variante I1b1b (M26)) e I1b2 che raggiunge discrete frequenze lungo le coste nord-occidentali dell'Europa continentale e in Sardegna. Dentro I1b2, si è formato I-M284 in Europa nord-occidentale e Isole Britanniche.
Aplogruppo R
Tutti gli aplotipi afferenti all'aplogruppo R condividono le mutazioni M207 (UTY2), M306 (S1), S4, S8, S9 e possono essere suddivisi in due principali linee evolutive: R1a e R1b.
La R1a potrebbe essersi originata nelle steppe euroasiatiche a nord del Mar Caspio e del Mar Nero. È associato alla cultura Kurgan, nota per la domesticazione del cavallo (circa 5000 anni fa). Questa linea è attualmente presente in Asia centrale e occidentale, India, e nelle popolazioni slave dell'Europa orientale.
La linea R1b è la più comune nelle popolazioni europee. Nell' Irlanda occidentale raggiunge una frequenza prossima al 100%. Si è originata prima della fine dell'ultima glaciazione e si è concentrata nei rifugi del sud-Europa per poi riespandersi verso nord con il progressivo mitigarsi del clima a partire da 14.000 anni fa.
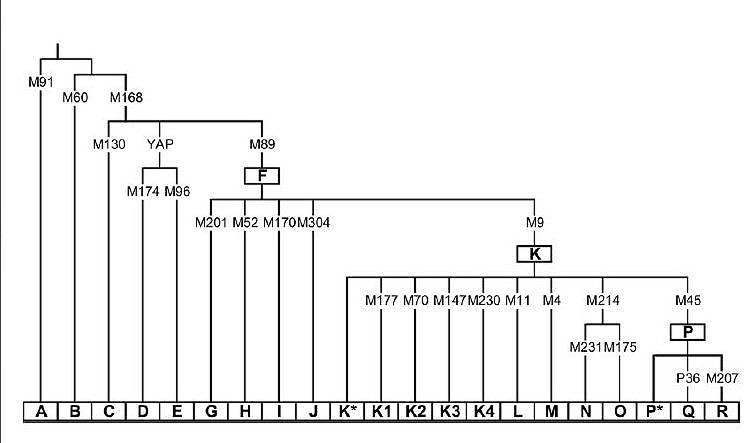
Riepilogo
delle mutazioni
che determinano i principali aplogruppi del cromosoma Y umano
Ipotesi
genetiche
sul popolamento dell'Europa
Le ipotesi genetiche sul popolamento dell'Europa sono un'insieme di teorie e di rilevazioni scientifiche che si propongono di contribuire alla ricostruzione delle vicende del popolamento del continente europeo a partire dall'analisi genetica dei suoi abitanti attuali. Si tratta di un campo di studi ancora in via di elaborazione, pertanto pochi sono i risultati considerati certi e definitivamente acquisiti dalla ricerca scientifica.
Gli studi si concentrano sull'analisi degli aplogruppi del cromosoma Y; a riguardare specificamente l'Europa sono l'aplogruppo G (maggiori incidenze: Ossezia, Georgia, Medio Oriente e Sardegna), l'aplogruppo I (maggiori incidenze: Scandinavia, Sardegna e Balcani) e l'aplogruppo R (maggiori incidenze: Europa centro-orientale e Isole britanniche).
L'origine degli aplogruppi più diffusi in Europa
In considerazione della maggiore o minore antichità di un aplogruppo, e delle sue incidenze nelle varie aree del mondo, sono state stabilite correlazioni tra gli aplogruppi e determinate culture storiche. Per quel che riguarda l'Europa, l'aplogruppo G, originato in Medio-Oriente, nel Caucaso o forse nell'odierno Pakistan, intorno a 30.000 anni fa, secondo alcuni studi potrebbe essersi diffuso in Europa nel Neolitico, oppure, vista la sua forte discontinuità, aver raggiunto l'Europa già nel Paleolitico.
L'aplogruppo I si ritiene si sia originato in Europa prima dell'ultima glaciazione. È probabile che sia stato confinato nel rifugio balcanico durante la glaciazione e che poi si sia ridiffuso verso nord con il ritiro dei ghiacciai. All'aplogruppo appartengono due subcladi, ognuna delle quali marca un'indipendente espansione della popolazione lungo diversi percorsi migratori durante e immediatamente dopo la glaciazione: l'I1a registra le più alte frequenze in Scandinavia, Islanda ed Europa nord- orientale (tanto che una sua mutazione, ricorrente sulle Isole britanniche, è interpretata come marcatore di invasioni scandinave).
L'aplogruppo R si ripartisce in due principali linee evolutive. La prima potrebbe essersi originata nelle steppe euroasiatiche a Nord del Mar Caspio e del Mar Nero ed è associata alla cultura Kurgan, nota per la domesticazione del cavallo (circa 5000 anni fa). La seconda è la più comune nelle popolazioni europee (in Irlanda occidentale raggiunge una frequenza prossima al 100%); si è originata prima della fine dell'ultima glaciazione e si è concentrata nei rifugi dell'Europa meridionale, per poi riespandersi verso Nord con il progressivo mitigarsi del clima (a partire da 14.000 anni fa).
L'aplogruppo R1a e gli Slavi
In Europa l'aplogruppo R1a si trova maggiormente in Polonia (circa il 60% della popolazione), in Serbia, in Croazia e in Bosnia (40-50%), nella Repubblica di Macedonia (35%) e nella Repubblica Ceca (32%). Si trova a circa il 15-25% nell'Europa settentrionale e al 45% in India secondo l'ipotesi di Kurgan. L'espansione potrebbe essere dovuta alle migrazioni indoeuropee verso i Balcani che seguirono l'Era glaciale. La distribuzione storica del geni può essere validata utilizzando il DNA, ma finora non ci sono dati riguardo all' aplogruppo Y dai corpi preservati nel ghiaccio di Ötzi o nella mummia di Taklamakan.
In Polonia il 60% dei polacchi possiede il R1a Y-DNA, mentre il 30% è composto dall'aplogruppo R1b e I1. La Germania è una nazione di DNA R1b, con una frequenza del R1a di circa l'8%. Questa chiara corrispondenza tra le lingue tedesche e quelle slave e la configurazione genetica R1a e R1b ha portato i ricercatori genetici a concludere che l'R1a è una valido identificatore dei popoli slavi, anche se questa teoria presenta non poche problematiche.
In contrasto alle credenze popolari, gli Svedesi e i Norvegesi non Slavi hanno una frequenza dell'R1a maggiore di quanto ci si possa aspettare (18% e 24% rispettivamente). Nel Regno Unito la distribuzione del R1a corrisponde a quella delle aree di influenza vichinga. Lo stesso aplogruppo Y significa che i vichinghi sono legati geneticamente a Ludzie, dove la concentrazione del R1a è la più alta in Europa.
Sia in Germania che in Polonia R1b e I1 esistono in proporzione 55%-40% rispettivamente, e sono gli unici paesi nordici ad avere simili proporzioni tra l'R1b e l'I1. Curiosamente, secondo la teoria autoctona, gli slavi occidentali della Germania e della Polonia non sono generalmente riconosciuti come popoli pre-slavi che divennero poi slavizzati. Gli archeologi polacchi additano anche alla cultura della Pomerania come la sorgente della distinzione culturale degli slavi in Europa. Questa era una forma di cultura lusaziana con grandi influenze vichinghe.
Fu la cultura degli Slavi occidentali a imporsi su quella degli Slavi orientali e meridionali, e ciò portò alla lechitizzazione da parte degli slavi occidentali, i fondatori della civilizzazione dell'Europa orientale. Le distinzioni culturali che appaiono ancora oggi negli abitanti della Rutenia (slavi orientali) è quindi ciò che resta della cultura originale slava.
Potrebbe essere vero che la divisione R1b/R1a risalga a prima dell'attuale confine tra le aree di lingua slava e lingua germanica, e che l'R1a non sia poi così indicativo delle persone slave come si presumeva inizialmente basandosi sulla divisione Polonia-Germania. Potrebbe però indicare, almeno nell'Europa settentrionale, un iniziale movimento est-ovest dei popoli che entrarono in Scandinavia ma furono per qualche ragione incapaci di penetrare nel cuore dell'Europa attraverso la Germania.
Gli esempi Sorbi hanno il 63% dell'Y-DNA R1a, e sono considerati almeno parzialmente slavi del sud.
I Venedi sono esempi di slavi occidentali puri, e sono considerati come popoli proto-germanici e proto-slavi: gli scienziati li considerano come i portatori del YDNA I1a in Europa principalmente tra gli Slavi, tra i Germani del nord e i Sardi. I Venedi sono anche menzionati come una delle tre divisioni dei popoli slavi del VI secolo in Europa dai giordani.
L'aplogruppo I (Y-DNA) (M170 Y-DNA) che in Polonia e Germania (il secondo paese che storicamente contiene la più grande popolazione di Slavi occidentali oltre la Polonia) è l'unico DNA che può essere associato ai Lechiti (23%), che sono slavi occidentali e si trova a più del 20% in tutti i Paesi slavi con la rara eccezione della Russia. I nativi esistono solo in Europa e il gene è associato agli antichi "barbari del nord". La grande maggioranza dei cromosomi Y in questi stati "barbari del nord" è comunque compreso negli stessi aplogruppi (il tedesco R1b nell'Europa occidentale, con l'eccezione dell'Italia, della Svizzera e della Penisola Iberica; negli Slavi orientali l'R1a nell'Europa orientale eccetto la Russia e N nell'Europa settentrionale a eccezione della Finlandia).
Nonostante i risultati delle ultime ricerche genetiche siano ancora provvisorie si diffonde sempre più tra gli studiosi la tesi inizialmente propugnata da pochi storici di una antica origine autoctona dei Celti risalente al mesolitico.
Il responsabile della diffusione di questa tesi è l'aplogruppo R1b del cromosoma Y; gli aplogruppi possono essere immaginati come i grandi rami dell'albero genealogico della componente maschile della specie Homo sapiens. Questi rami-aplogruppi mostrano come si sono sviluppate le popolazioni e ne definiscono perciò anche l'ambito geografico di sviluppo.
Nel continente europeo sono diffusi 4 aplogruppi: E, J, I e R che è quello che più interessa analizzare e che si divide in due sottogruppi (o aplotipi) R1b nelle popolazioni sud-occidentali e R1a in quelle dell'Europa orientale.
L'aplogruppo
R1b, nella sua mutazione M343, compare in Europa già 30.000 anni fa con l'
uomo di Cro-Magnon, del genere Homo sapiens e diretto progenitori degli
attuali Europei, ma si attesta verosimilmente solo dopo l'ultima era glaciale.
Le moderne popolazioni celtiche atlantiche sono contraddistinte dall'aplotipo
R1b, ma R1b non è esclusivo di queste popolazioni: l'aplotipo R1b
caratterizzato dai marcatori SNP e STR è presente in tutta la Spagna, Baschi,
Galles e Irlanda ma ha alte frequenze anche in Sardegna, Corsica e Toscana. Il
collegamento tra popolazioni della Spagna e delle Isole britanniche potrebbe
essere dovuto a migrazioni di genti mesolitiche (8000 - 5000 a.C.) dalla
Penisola Iberica, che con il mitigarsi del clima si spostavano dai rifugi
glaciali per ricolonizzare il nord dell'Europa. Nel resto d'Europa l'aplotipo
R1b è presente in maniera degradante da ovest verso est dove praticamente
scompare per lasciare il posto all'aplotipo R1a a partire da un confine
naturale che va dalle Alpi orientali al mar baltico.
È senz'altro molto rilevante notare il fatto che l'epicentro della diffusione della cultura celtica abbia basse frequenze di R1b: ciò dimostrerebbe che il panorama genetico attuale sia sostanzialmente quello disegnato nel periodo mesolitico e che la diffusione della cultura celta sia stata più di tipo culturale (ad esempio imposto da classi dominanti celte) che dovuto alla diffusione di persone, e quindi di geni.
Da segnalare anche una massiccia presenza dell'aplotipo R1b anche nell'Italia del nord, non è ancora chiaro però se la sua presenza sia dovuta alle migrazioni iniziali del paleolitico provenienti da una regione imprecisata dell'Asia occidentale o a eventi più recenti legati all'ultima glaciazione (tra i 15.000 e i 10.000 anni fa), durante la quale i contatti tra popolazioni iberiche e quelle italiche erano consentiti e facilitati dal congelamento di ampi tratti del Mediterraneo settentrionale che formavano in questo modo ponti naturali facilmente percorribili. A ogni modo, poiché l'ultima glaciazione rese inabitabili le regioni settentrionali e centrali dell'Europa, è ovvio che l'apparizione aplogruppo R1b in Italia preceda quella nelle isole britanniche.
Da queste poche evidenze si deduce che l'aplotipo R1b presente nelle moderne popolazioni celtiche e già presente nelle popolazioni del mesolitico, non può essere stato introdotto da migrazioni di popoli indoeuropei in epoca più recente, attestando così, secondo i dati biologici, l'originalità e l'europeicità dei Celti di ogni epoca.
Questa prospettiva autoctona è compatibile con la teoria della continuità proposta da Colin Renfrew, ma tuttavia viene generalmente rigettata dagli studiosi di linguistica, che da più di un secolo si sono posti il problema dell'origine dei Popoli Celti, e per un lungo periodo del secolo scorso furono gli unici studiosi a presentare ipotesi, frutto delle loro ricerche, riguardo tale origine. La loro obiezione è imperniata sulla constatazione delle strette prossimità dialettali tra le varie lingue celtiche: se si fossero effettivamente sviluppate in un'area tanto vasta, per millenni, senza scrittura e in assenza di qualsiasi unità politica, avrebbero dovuto differenziarsi tra loro molto di più di quanto non sia storicamente attestato. La linguistica storica, quindi, indica un periodo di separazione rispetto alla lingua protoceltica di poche centinaia di anni.
Marcatori del cromosoma Y ereditati nella linea paterna consentono la ricostruzione della storia naturale sulla base delle mutazioni via via acquisite. La genetica della popolazione umana sarda consiste nello studio del pool genico degli attuali abitanti dell'isola con due principali obiettivi. Il primo ha uno scopo prettamente scientifico e culturale ed è quello di ricostruire la storia naturale della popolazione. Essa consiste nella comprensione dell'entità, dei tempi e delle modalità della fondazione unitamente alle successive o concomitanti dinamiche demografiche e evolutive. L'altro è invece applicativo e ha la finalità di comprendere le cause genetiche di alcune patologie sfruttando alcune peculiarità della popolazione sarda, che la rendono di elezione per studi che prevedono l'utilizzo di isolati genetici. Entrambi gli obiettivi sono perseguiti attraverso lo studio molecolare di marcatori del DNA di individui della popolazione mediante un approccio multidisciplinare che coinvolge biologi, medici, naturalisti, statistici, bioinformatici, biotecnologi, archeologi, antropologi e paleontologi.
Evoluzione e Demografia
Il numero di mutazioni accumulate in regioni cromosomiche non ricombinanti, è stato impiegato come orologio molecolare. L'interpretazione dei risultati farebbe risalire in modo lineare la popolazione sarda attuale da una popolazione ancestrale fondatrice che potrebbe aver raggiunto l'isola in più ondate durante il Paleolitico superiore. I geni dei Sardi si inquadrano perfettamente nell'ambito del pool genico europeo con grosse differenze però in termini di:
Frequenze geniche (per lo più dovuti a effetto del fondatore e deriva genetica casuale). Ad esempio l'aplotipo I-M26 del cromosoma Y, presente anche nella penisola iberica (1-7%) ha in Sardegna frequenze fino oltre il 30%.
Presenza di sottotipi sardo-specifici (da imputarsi a mutazioni occorse nell'isola dato il lungo tempo intercorso dalla fondazione ad oggi). Ad esempio, l'aplotipo R-M18 è esclusivo della Sardegna e origina per acquisizione di una mutazione dall'aplogruppo più antico R-M173.
Dimensione della popolazione ancestrale
L'elevata variabilità genetica implica un numero rilevante di linee fondatrici. L'archeologia indica che la taglia effettiva della popolazione sarda sia stata molto importante relativamente alle altre aree geografiche coeve. Le grandi crisi demografiche medievali infatti non hanno potuto cancellare la struttura della popolazione come è successo ad esempio nella vicina Corsica.
Selezione
Le frequenze di alcuni geni sono stati influenzati dalla presenza della selezione operata dal plasmodium, l'agente infettivo della malaria che, durante il suo ciclo vitale parassitizza i globuli rossi del sangue dell'uomo e le ghiandole salivari della zanzara. La selezione ha agito aumentando la frequenza di geni che possono causare l'insorgenza di a- e ß-talassemie o del favismo attraverso un processo noto come polimorfismo bilanciato.
Struttura della popolazione
Presenza di alta variabilità interindividuale, ma bassa variabilità tra subpopolazioni geografiche o linguistiche. Questa assenza di substruttura rende la popolazione ottimale per lo studio e la comprensione delle cause genetiche di patologie a eziologia ancora ignota, soprattutto di quelle che presentano alta incidenza nell'isola, come le malattie autoimmuni diabete e sclerosi multipla. Infatti una casistica maggiore di individui affetti si può ottenere in una popolazione vasta e omogenea. Tale caratteristica della popolazione sarda non è però unanimemente accettata. Infatti, precedenti studi hanno evidenziato una elevata variabilità tra subpopolazioni geografiche o linguistiche, mentre l'intera popolazione sarda presenterebbe livelli di eterogeneità paragonabili a quelli delle popolazioni continentali, non rendendola uno strumento utile per lo studio delle malattie multifattoriali. Solo alcune sottopopolazioni (ad esempio l'Ogliastra) caratterizzate da un elevato grado di isolamento e di omogeneità, sarebbero proprie a questo scopo.
Flusso genico
L'omogeneità della popolazione contrasta con fenomeni di flusso genico localizzato (dalla Spagna, dall'Africa, ecc.). I dati genetici sono consistenti con un effetto assai blando in termini di flusso genetico operato dai popoli colonizzatori delle epoche neolitiche e storiche.
Malattie genetiche frequenti
Diabete di
tipo I
Malattia di Wilson
Sclerosi multipla
Thalassemia beta39
APECED
Sclerosi multipla
La sclerosi multipla OMIM#126200 è una patologia autoimmune che colpisce il sistema nervoso e che è diffusa soprattutto tra le popolazioni europoidi. La incidenza di questa patologia tra i Sardi è elevata e comparabile solo con quella delle popolazioni nord-europee. Questo contrasta con l'ipotesi di un cline di incidenza nord-sud in Europa che potrebbe essere correlato a un qualche fattore ambientale eziologico ancora sconosciuto. Inoltre, dall'analisi della ricorrenza della malattia nei fratelli di numerosi pazienti, unitamente ad altri elementi epidemiologici o di rischio quali il sesso, l'età di esordio, il luogo di nascita e la presenza di altri famigliari affetti oltre i fratelli, si è osservato un maggior rischio di incidenza nei fratelli dei pazienti rispetto alla popolazione generale. In aggiunta, per valutare la presenza di familiarità tra pazienti non imparentati, 11 individui provenienti da un singolo paese sono stati correlati genealogicamente tra loro mediante pedigree, rivelando che discendevano tutti da sole 3 coppie di antenati. In generale l'aumento dell'incidenza tra persone che condividono parte del patrimonio genetico starebbe a indicare un effetto genetico nella manifestazione della malattia. Queste evidenze suggeriscono che geni di suscettibilità alla sclerosi multipla fanno parte del pool genico dei Sardi sulla base dell'ipotesi di una condivisione di varianti genetiche per discesa da comuni progenitori.
Longevità
In Sardegna è presente un'intensa attività di ricerca sulla genetica che coinvolge la popolazione (caratterizzata da un patrimonio genetico omogeneo) e che riguarda sostanzialmente due progetti:
Progetto "ProgeNIA" - Utilizzo della popolazione sarda, per la sua omogeneità, per lo studio dei tratti fenotipici legati all'invecchiamento, e di malattie complesse;
Progetto "AKeA" - Studio dei marcatori della salute e della longevità dei Sardi.
Il Progetto "ProgeNIA", iniziato nel novembre del 2001, è nato dalla collaborazione tra il Consiglio Nazionale delle Ricerche, il National Institute of Aging degli Stati Uniti (che lo finanzia interamente) e il National Institute of Health (NIA-NIH); vede la partecipazione attiva degli abitanti di Lanusei, Ilbono, Arzana ed Elini (doveva concludersi entro il 2005, ma è stato rifinanziato fino al 2011).
"Gli studi del Progetto Progenia ... mirano a identificare i geni e i fattori ambientali responsabili dell'invecchiamento dell'uomo." e hanno portato alla scoperta dell'esistenza di "una relazione tra adiposità, circonferenza della vita e quantità di insulina presente nell’individuo. Poi è stato individuato il rapporto, nella formazione del colesterolo, tra ereditarietà e ambiente (il cibo e lo stile di vita): il 40 per cento la prima e il 60 il secondo. E ancora: si è rilevato che negli anziani longevi l’ereditarietà ha più peso nelle donne che negli uomini; e che, a seconda di come si combina una serie di caratteristiche di vita e di stato di salute, l’influenza dei geni si fa sentire più o meno tardivamente.".
Sulla rivista scientifica Plos Genetics sono stati pubblicati i risultati ottenuti dagli studi condotti su ereditarietà delle caratteristiche cardiovascolari e di tratti della personalità su 6148 sardi e sui geni responsabili dell'obesità. Oltre alle scoperte sull'obesità, gli studi sui volontari che partecipano al progetto Progenia hanno portato alla scoperta del gene responsabile della variazione dell'acido urico, di un gene responsabile per l'altezza e di 7 nuovi geni del colesterolo.
Il progetto "AKeA" (acronimo di "A Kent’Annos", tradizionale augurio sardo che significa "a cent'anni"), basato sugli studi effettuati a partire dal 1997 dal team del Prof. Luca Deiana e presentato ufficialmente nel febbraio 2002, fa capo alla cattedra di Biochimica Clinica dell’Università di Sassari e vede la collaborazione del Max-Planck Institute for Demographic Research, Rostock, Germania e dalla Duke University, North Carolina, USA. Il monitoraggio viene fatto su tutta la popolazione sarda.
I risultati dello studio "AKeA" sono stati pubblicati e presentati in vari simposi internazionali che hanno visto la partecipazione di ricercatori di tutto il mondo, giunti nell'isola per studiare il DNA dei Sardi, ed hanno suscitato anche l'interesse dei mass-media.
Nella Sardegna, infatti, è stata constatata la presenza di numerosi ultracentenari (nel luglio 2007 sono più di 330), in media circa 22 ogni centomila abitanti, contro una media tra gli 8 e i 10 in altre parti del mondo. Questo rapporto risulta in crescita col passare del tempo, dal momento che nel periodo '97-'99 la media era di 13,5 ultracentenari, e nel 2000 era salita a 19.
Sono state individuate delle zone interne ad alta concentrazione di longevi, e si è scoperto che il rapporto maschi/femmine ultracentenari in Sardegna è ben diverso da quello presente altrove. Se nel resto d'Italia e in Occidente il rapporto è di 1 a 4, se non addirittura di 1 a 7, nell'Isola è generalmente al di sotto di 1 a 2, per diventare paritetico nelle aree interne.
Sono state formulate molte spiegazioni di questa particolarità, come la qualità della vita o un particolare regime alimentare, ma principalmente gli studiosi sono interessati ad analizzare specifici fattori genetici che interagirebbero in concomitanza con i fattori ambientali.
L'Isola vanta alcuni primati:
L'uomo più vecchio del mondo, attestato dal Guinness dei Primati 2001. Antonio Todde (chiamato Tziu Antoni), nato a Tiana (NU) il 22 gennaio 1889, è scomparso il 3 gennaio del 2002, poche settimane prima del compimento del 113° compleanno; attribuiva la sua longevità al bicchiere di buon vino rosso che beveva ogni giorno.
L'uomo più vecchio d'Europa, e al terzo posto in tutto il mondo, nel 2003. Giovanni Frau, nato a Orroli (NU) il 29 dicembre 1890, è scomparso il 19/06/2003 all'età di 112 anni.