Vol. 2° - III.4.
IL NUCLEOSOMA
Per nucleosoma s’intende una struttura formata dal cilindretto di istoni, o core, alla cui superficie si avvolge il DNA.
Quindi, il nucleosoma è la subunità elementare della cromatina privata delle proteine non istoniche. DNA e istoni sono organizzati in unità dette nucleosomi, costituiti da un nucleo centrale discoide con 8 molecole di istoni denominato ottamero di istoni, a forma di limone; ad avvolgere tale nucleo si trova il DNA che forma 2 giri completi per ciascun nucleosoma.
Ogni nucleosoma giace vicino all’altro, ed è collegato all’altro dal filamento continuo di DNA strutturato come un solenoide. I nucleosomi sono inoltre strettamente saldati tra loro da un’ulteriore molecola di istone, l’istone H1.
Il superavvolgimento di DNA permette una struttura
compatta dei geni, come si verifica per l’eucromatina, nonché una loro
ulteriore compattazione, caratteristica dell’eterocromatina.
I tratti di DNA lunghi circa 60 paia di basi (60 bp) che collegano due nucleosomi tra loro sono detti spacer, o meglio, linker, tratti spaziatori o di unione.
Il componente geneticamente più importante della cromatina è il DNA, in quanto a livello di questa molecola avviene la trascrizione degli RNA e, nella fase S del ciclo cellulare, si attua la duplicazione del DNA stesso. Pare che gli istoni abbiano un ruolo strutturale nell’architettura della cromatina entrando nella costituzione dei nucleosomi e, almeno in parte, regolano anche l’attività del DNA; questo vale specialmente per l’istone H1.
Si ritiene però che nella regolazione del DNA
abbiano importanza maggiore le proteine non-istoniche, che appartengono a una
classe molecolare molto più ricca e più varia rispetto ai pochi tipi di
istoni conosciuti. Queste proteine, che aumentano nella cromatina quando
questa è attiva, potrebbero agire selettivamente scegliendo i siti da attivare.
Gli istoni bloccherebbero, con le loro cariche basiche,
quelle acide del DNA, impedendo alla doppia elica di svolgersi e quindi di
trascrivere. Le proteine acide rimuoverebbero il blocco istonico in quanto,
neutralizzando le cariche degli istoni in porzioni specifiche del genoma,
permetterebbero al DNA di svolgersi localmente e di trascrivere.
4.1. DNA a sequenza unica
Geni strutturali
Il DNA della cromatina può essere suddiviso in unità funzionali: i geni.
Un gene è un tratto di DNA in grado di codificare, e di trascrivere in fase G1, una molecola di RNA. Il nome di gene per antonomasia spetta a quel tratto di DNA che codifica per una proteina, attraverso la trascrizione del relativo mRNA.
I geni delle proteine sono anche detti geni strutturali, che negli eucarioti sono molto più lunghi degli mRNA citoplasmatici da essi trascritti.
Altri tipi di geni sono i geni ribosomiali o nucleolari che trascrivono gli rRNA, e i geni transfer che codificano per i
tRNA. Talora a questi geni si dà rispettivamente il nome di rDNA e di tDNA.
Il genoma degli eucarioti contiene molti più geni
strutturali rispetto ai procarioti, in quanto sono almeno 10 volte più
numerosi. Si calcola che una cellula eucariotica sia in grado di sintetizzare
da 10.000 a 30.000 proteine diverse. Il DNA di questi geni, normalmente, è
presente in copia unica nel genoma aploide di una cellula, ove costituisce la frazione di DNA a copia singola.
Alcuni geni strutturali vengono ripetuti poche volte, e tutti lo sono almeno
due volte in una cellula somatica diploide.
4.2. DNA a sequenze ripetute
4.2. a. Frazione mediamente ripetitiva del DNA
Geni ribosomiali e transfer
Gli eucarioti presentano alcune centinaia di copie
dei geni per gli rRNA e per i tRNA,
costituendo parte della frazione
di DNA mediamente ripetitiva, in quanto costituita da
sequenze nucleotidiche eguali fra loro e ripetute da alcune centinaia ad
alcune migliaia di volte. Fanno parte di questa frazione anche i geni degli
istoni e i geni delle immunoglobuline; altre sequenze della stessa frazione
non trascrivono RNA citoplasmatici, ma forse RNA di controllo sull’attività
del genoma: si tratta dei geni
regolatori.
4.2.b. Frazione altamente ripetitiva del DNA
Una terza frazione del DNA
eucariotico, talora molto abbondante, è detta frazione altamente ripetitiva, perché costituita da brevi
sequenze nucleotidiche fra loro uguali e ripetute centinaia di migliaia,
talora milioni di volte. In alcuni casi questa frazione può essere isolata
dal resto del DNA, costituendo il cosiddetto
DNA satellite.
Il DNA satellite viene così distinto a seconda del numero
di nucleotidi costituenti le regioni ripetute:
·
macrosatelliti: regioni ripetute della
lunghezza di circa 100 nucleotidi
·
microsatelliti: le regioni ripetute
sono costituite da 2-6 nucleotidi.
Il DNA altamente ripetitivo non trascrive RNA; le sue funzioni sono ancora sconosciute, ma si
presume siano importanti nell’economia del nucleo. Infatti, pur essendo
questo DNA molto variabile da una specie all’altra, è tuttavia presente nel
genoma di quasi tutti gli eucarioti, e in taluni è talmente abbondante da
essere la componente principale della cromatina.
Al contrario di quanto si osserva nei procarioti, i geni
degli eucarioti sono distanziati fra loro da tratti di DNA senza apparente
funzione; tratti di DNA non trascritti in RNA citoplasmatici si ritrovano,
sempre negli eucarioti, addirittura all’interno dei geni strutturali.
Tra i geni, forse il più indagato è quello dell’ovalbumina, proteina dell’albume dell’uovo
di pollo secreta dalle cellule dell’ovidutto, costituita da circa 600
aminoacidi. L’mRNA citoplasmatico addetto alla sua codifica consta quindi di
1.800 nucleotidi (600x3, data la corrispondenza, o codice genetico, fra una
tripletta di nucleotidi e l’aminoacido codificato). La lunghezza di questo
mRNA è quindi pari a 1,8 kb (kilobasi o migliaia di nucleotidi). Con tecniche
di ibridazione molecolare si è visto che il gene strutturale dell’ovalbumina
è più lungo del suo messaggero citoplasmatico, in quanto è composto da
circa 7,7 kb.
Quindi, solo
una frazione del DNA eucariotico è addetta alla codifica delle proteine
cellulari, e talora tale frazione è
inferiore al 50%, mentre nei procarioti quasi tutto il DNA ha questa funzione.
Le funzioni del rimanente DNA, mediamente o altamente ripetuto, sono in gran
parte ancora da chiarire. Lungo la cromatina, tratti di DNA a copia singola
lunghi meno di 2 kb, si alternano regolarmente a tratti di DNA ripetitivo più
lunghi. Questo caratteristico pattern di interspersione fra le due classi di DNA sembra
presente nel genoma della maggior parte degli eucarioti.
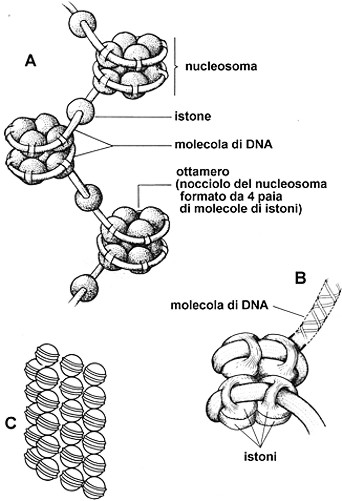
4.3. Struttura dei nucleosomi
|
|
Fig.
III. 13
- Struttura della fibra cromatinica. A
- fibra cromatinica a collana di perle. B
- Nucleosoma. che rappresenta la forma condensata della fibra cromatinica.
|
L’esame al microscopio elettronico delle nucleoproteine
estratte dal nucleo interfasico ha permesso, in certi casi, di verificare che
le fibre cromatiniche hanno una struttura a filo di
perle.
Ogni sferetta, detta nucleosoma, ha un diametro di circa 10 nm, ed è connessa
a quelle adiacenti da un filamento lungo 14 nm dello spessore di 3 nm. Il
nucleosoma presenta le seguenti caratteristiche:
§
possiede
un core proteico
formato da istoni (H2A, H2B, H3, H4) organizzati in ottamero (4 coppie, una
per ogni tipo di istone)
§
il
core è circondato da un segmento
di DNA avvolto a elica; la lunghezza di questo DNA corrisponde a 140
coppie di basi complementari
Un istone H1, situato all’esterno di ogni nucleosoma,
controllerebbe il grado di avvolgimento, o condensazione,
del filamento di cromatina. Il DNA compreso fra due nucleosomi contiene in
media 40 coppie di basi. Quando i nucleosomi sono accostati l’uno all’altro,
la fibra cromatinica ha un diametro di 10 nm. Questa fibra può spiralizzarsi
assumendo la forma di una fibra del diametro di 20 nm.
4.4. Aspetti della fibra cromatinica
4.4.a. Forma decondensata
La fibra ha l’aspetto di una collana
di perle con i nucleosomi distanziati fra loro. Quando il DNA
viene trascritto, il filamento nucleosomico si modifica: gli ottameri si
separano in due tetrameri in modo che il filamento diventi eminucleosomico. La
replicazione comporta la demolizione del nucleofilamento che si autoduplica e
un nuovo assemblaggio di istoni per ognuna delle due molecole figlie di DNA.
4.4.b. Forma condensata
L’istone H1 può imporre al
nucleofilamento o fibra nucleosomica, detta anche fibrilla di cromatina, una
superspiralizzazione che dà origine a fibre lunghe 10 nm. La fibra di
cromatina corrisponde a una superspiralizzazione del nucleofilamento che forma
un solenoide, reso stabile dall’interposizione
degli istoni H1 entro ogni giro di spira. Il diametro è compreso fra 20 e 25
nm.
4.4. c. Geni repressi, base della differenziazione cellulare
4.4.c.1. Eterocromatina
L’eterocromatina è costituita
da nucleosomi maggiormente ravvicinati e avvolti ad elica. Essa corrisponde
all’80% del DNA nucleare e si presenta sotto forma di zolle molto
elettrondense. Si tratta di segmenti di cromosomi
che rimangono condensati dopo la mitosi e sono porzioni di DNA geneticamente inattive,
in quanto non vengono trascritte. L’eterocromatina ha la superstruttura a
solenoide, contiene poco RNA e non incorpora l’uridina triziata; è molto
ricca in istoni di tipo H1 ed esiste sotto due forme:
q Eterocromatina
costitutiva: è una struttura ripetitiva, priva di valore
genico, che quindi non codifica;
rappresenta segmenti identici di cromosomi che conservano, durante tutto il
ciclo cellulare, un aspetto condensato (per es., il braccio lungo del
cromosoma Y umano).
q
Eterocromatina
facoltativa: è così denominata perché in particolari condizioni
sia naturali che sperimentali è in grado di trasformarsi in eucromatina,
ossia in cromatina attiva nella trascrizione di RNA; nell’eterocromatina
facoltativa i geni
sono repressi; questa completa inattivazione di un segmento di
cromosoma non si accompagna a un’alterazione della struttura genica. Le
sequenze di geni repressi sono diverse da un tipo cellulare all’altro e, a
seconda del tipo cellulare, i geni che si esprimono non sono sempre gli
stessi: questo meccanismo sta alla base
della differenziazione cellulare.
4.4.c.2. Eucromatina
L’eucromatina nelle zone,
dette in microscopia ottica
intercromatiniche, le fibre cromatiniche hanno la forma di filamenti
nucleosomici e sono geneticamente attive. Questa eucromatina,
formata da DNA non ripetitivo, è sede di processi trascrizionali per la
sintesi di RNA messaggero (mRNA) e di trasferimento (tRNA).
4.5. Struttura del DNA
Gli acidi nucleici rappresentano le molecole più grandi e più
interessanti presenti negli organismi viventi. Le informazioni necessarie per
costruire un intero organismo e per mantenere in vita ciascuna cellula
mediante la sintesi di tutte quante le proteine cellulari, sia strutturali che
funzionali, vengono codificate in una macromolecola che fa parte degli acidi
nucleici, anch’essi costituenti organici della sostanza vivente.
Fig. III. 14 - Simbolo dell’Ariete. L’Ariete codifica gli inizi, quindi le molecole che danno origine alla vita e che si organizzano in sistemi. Pertanto governa il DNA, il sangue, il sistema immunitario e le difese dell’organismo.
Quando furono isolati, fu subito evidente che gli acidi
nucleici si presentavano diversi dalle proteine e dagli altri componenti dell’organismo:
erano di natura acida ed erano ricchi di fosforo. Poiché furono isolati dal
nucleo delle cellule, venne loro attribuito l’aggettivo
nucleici. Solo successivamente fu osservata la loro presenza anche nel
citoplasma.
Esistono
due tipi di acidi nucleici la cui composizione chimica è molto simile:
·
DNA «
acido desossiribonucleico
·
RNA «
acido ribonucleico
Essi sono sempre presenti in
ogni forma di vita, sia nelle cellule procariotiche che in quelle
eucariotiche. Nei virus è presente soltanto DNA oppure RNA. Gli acidi
nucleici sono dei polinucleotidi, in
quanto la loro molecola è formata dall’unione di numerosissime unità
semplici, i nucleotidi. Negli acidi
nucleici i nucleotidi formano dei polimeri
dando luogo a catene la cui struttura può variare in base alla sequenza
nucleotidica.
Fig. III. 15 - DNA mentre si apre per la trascrizione. L’apertura simbolizza l’avvio dei meccanismi necessari alla sintesi delle molecole vitali.
L’unione tra due nucleotidi è resa possibile dal fatto
che l’acido ortofosforico impegna due dei suoi tre radicali acidi nell’esterificare
il gruppo alcolico in posizione 3 di una molecola di zucchero e in posizione 5
della molecola di pentoso successiva: si formano così catene in cui i vari
nucleotidi sono uniti da esterificazione
fosforica tra il radicale OH del carbonio 5 di uno zucchero e l’OH
del carbonio 3 di uno zucchero successivo e così via. Queste molecole
polinucleotidiche sono acide perché ogni residuo fosforico ha ancora un
radicale OH libero.
Fig. III. 16 - Molecola di anticorpo. La forma ripete quella dell’Ariete.
4.6. I mononucleotidi del DNA e dell’RNA
Questi monomeri contengono, in
quantità equimolecolare, l’acido fosforico, uno zucchero a 5 atomi di
carbonio, detto pentoso, e una base
azotata a struttura ciclica.
Il composto formato da zucchero + base azotata prende il
nome di nucleoside.
Così, una molecola di DNA è formata dall’unione di una molecola di acido
fosforico con un nucleoside.
q
acido ortofosforico
H3PO4 con 3 funzioni acide; due si legano al carbonio delle
molecole di zucchero
q
pentoso: zucchero a 5 atoni di
carbonio, desossiribosio per il DNA, ribosio
per l’RNA
q
basi azotate a struttura ciclica:
le basi azotate del DNA e dell’RNA derivano da composti aromatici
eterociclici, purina e pirimidina:
q basi puriniche, identiche nel DNA e nell’RNA:
o
Adenina
« A
o Guanina
« G
q basi pirimidiniche, diverse per DNA e RNA, con citosina in comune
q per il DNA
o Timina
« T
o
Citosina
« C
q per l’RNA
o Uracile
« U
I desossiribonucleotidi e i ribonucleotidi sono gli unici costituenti base della molecola di DNA e di RNA.
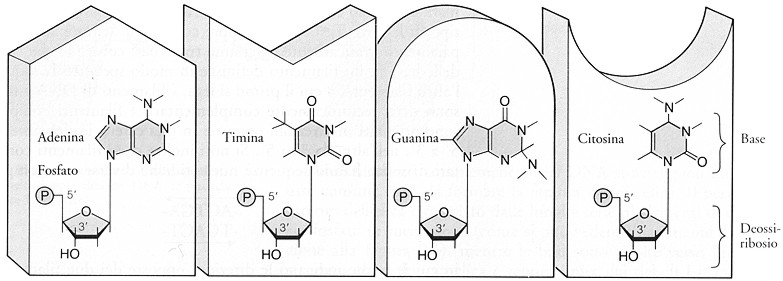
Fig. III. 17 - I nucleotidi del DNA. Le superfici superiori a incastro vogliono indicare l’appaiamento esclusivo e complementare delle molecole tra loro.
4.7. Struttura primaria del DNA
4.7.a. Filamento di DNA o molecola monocatenaria
Il DNA è un polimero
lineare di mononucleotidi. I quattro tipi di desossiribonucleotidi vengono
uniti tra loro dal legame teso fra acido fosforico e desossiribosio di due
nucleotidi vicini. Il radicale fosforico unisce il carbonio 3’ di un
desossiribosio al carbonio 5’ di un desossiribosio adiacente per mezzo di un
legame 3’-5’ fosfodiesterico. Lateralmente, sul carbonio 1’ di ciascun
desossiribosio, si lega una base purinica o pirimidinica. I
desossiribonucleotidi possono raggiungere un numero pari a 104¸105
per filamento.
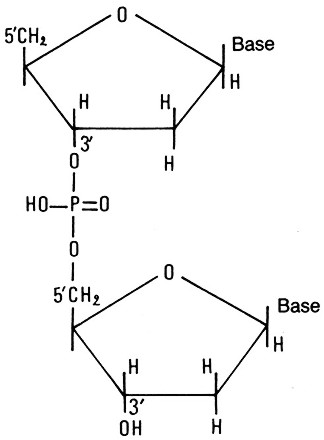
Fig. III. 18 - Legame fosfodiesterico teso fra le basi azotate
4.7.b. Molecola di DNA
È costituita da due filamenti
tenuti insieme da legami a idrogeno, i quali uniscono le basi puriniche e
pirimidiniche. Si tratta di un appaiamento
esclusivo come conseguenza della forma delle molecole: è
necessario che l’appaiamento sia complementare (complementarietà sterica) affinché avvenga l’associazione. Così
la molecola di DNA viene ad assumere la
forma di una scala in cui
·
i montanti sono costituiti dalla
successione: 3’ §
zucchero §
5’ Õ
PO4 Ö
3’ § zucchero § 5’
·
i pioli risultano dall’insieme:
zucchero Ö
base x
base Õ
zucchero.
4.8. Struttura tridimensionale del DNA
La molecola di DNA è costituita da 2 filamenti elicoidali antiparalleli, avvolti intorno a un asse comune a formare una doppia elica.
Il passo della doppia elica è lungo 3,4 nm, contiene dieci paia di desossiribonucleotidi distanziati di 0,34 nm.
La doppia elica ha un diametro di 2 nm.
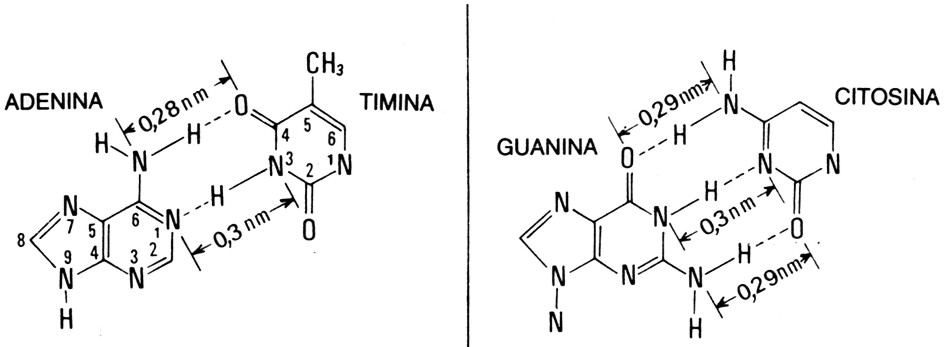
Fig. III. 19 - Legami a idrogeno che uniscono i filamenti di DNA
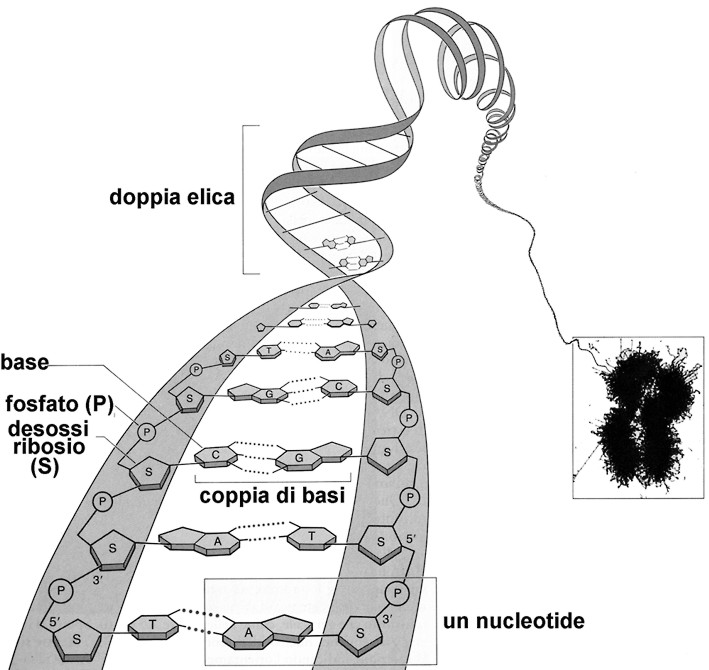
Fig. III. 20 - Organizzazione e associazione dei nucleotidi nella doppia elica del DNA
La
lunghezza della molecola dipende dalla cellula alla quale appartiene. Il
DNA dell’Escherichia coli è lungo
1 mm e contiene 4x106
paia di desossiribonucleotidi, mentre il DNA di una cellula umana aploide
(come lo spermatozoo) ne contiene 109
e, se misuriamo il DNA disteso dei 23 cromosomi, avrebbe una lunghezza totale
di 1 m.
Il termine antiparallelo
deriva dal fatto che lo scheletro di uno dei due filamenti ha questa sequenza:
·
fosfato
(PO4)
·
carbonio
3’ del desossiribosio
·
carbonio
5’ del desossiribosio
·
fosfato,
ecc…
PO4 Ö
3’ « 5’ Õ PO4
Ö 3’ « 5’ Õ PO4
invece l’altro filamento ha
una sequenza opposta al primo, cioè:
PO4 Ö
5’ « 3’ Õ PO4
Ö 5’ « 3’ Õ PO4
Nella doppia elica i due filamenti che si avvolgono in giri destrorsi cono costituiti da catene zucchero+fosfato orientate in direzioni opposte; tali filamenti esterni sono detti scheletri e costituiscono la porzione invariabile del DNA.
I filamenti antiparalleli sono mantenuti a regolare distanza fra loro (2 nm) da basi azotate che sporgono dagli scheletri verso il centro della doppia elica. Tali basi, disposte in una fila per scheletro, si collegano fra loro con legami a idrogeno, mantenendo quindi unite le due metà di DNA. La rottura di questi legami, che si verifica in certe fasi dell’attività cellulare, permette ai due filamenti di DNA di separarsi e ciascuno può funzionare da stampo per la sintesi di nuovi polinucleotidi.
Per motivi stereochimici, al
centro della doppia elica la base A può legarsi solo con la T e la base C
solo con G: in tal modo il diametro di 2 nm del DNA si mantiene regolare.
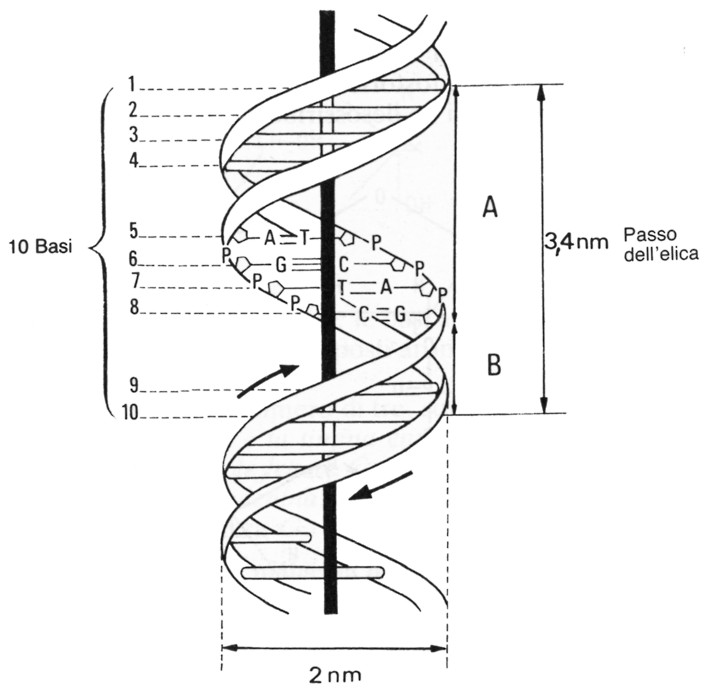
Fig. III. 21 - Struttura terziaria del DNA
L’appaiamento obbligatorio di A con T e di C con G rende i due filamenti fra loro complementari: se uno di essi presenta la sequenza C-T-A-C-G, l’altro non potrà che avere la sequenza complementare G-A-T-G-C. Un filamento costituisce quindi una sorta di stampo per l’altro: la doppia elica, aprendosi, separa i due vecchi filamenti, ciascuno dei quali funge da stampo per la sintesi di un nuovo filamento, che risulta identico a quello vecchio complementare.
In tal modo nascono due molecole di
DNA uguali fra loro e uguali alla molecola di partenza, costituite ciascuna da
un nuovo e da un vecchio filamento. Questo meccanismo di autoduplicazione è
di tipo semiconservativo in quanto il nuovo DNA
è costituito per metà dal precedente.
Il DNA è
l’unica molecola dell’organismo in grado di duplicare se stessa
con l’aiuto delle DNA polimerasi e, poiché può raggiungere enormi
lunghezze, in un codice costituito dalla sequenza di basi lungo il filamento,
si presta molto bene a trasmettere un’enorme quantità di informazioni
genetiche. Per esprimere tali informazioni il DNA si serve di molecole che
sono una sua copia complementare: gli RNA.
Nei procarioti il DNA si presenta sotto forma di molecole
circolari, ripiegate ad ansa, probabilmente tenute insieme da molecole
ribonucleoproteiche. Nel nucleo degli eucarioti il DNA è sempre associato a
proteine, tra le quali le più stabili sono gli istoni, e con le quali forma
strutture caratteristiche granulari: i nucleosomi.
4.9. Quantità del DNA genomico
La quantità di DNA è costante nei nuclei somatici di ciascuna specie, mentre può differire da una specie all’altra. Ovviamente la costanza del DNA può essere controllata solo misurando nuclei allo stesso stadio del ciclo cellulare: abitualmente vengono presi in esame i nuclei nella fase G1 o presintetica.
Durante la fase G1 i nuclei somatici delle cellule umane hanno
una quantità di DNA pari a circa 7 pg (picogrammi
o miliardesimi di milligrammo), mentre nella rana la quantità è pari a 14 pg.
Nei nuclei presintetici di alcune classi animali la quantità di DNA è relativamente stabile: nei mammiferi si aggira sui 7 pg, negli uccelli fra 3 e 4 pg. Invece altre classi presentano specie con quantità di DNA molto diverse fra loro: tra gli anfibi, certe rane del deserto hanno 2 pg di DNA per nucleo, mentre l’Amphiuma, un urodelo a metamorfosi incompleta, supera i 150 pg.
Anche nel regno vegetale troviamo gruppi sistematici con quantità costante di
DNA e altri con quantità molto variabili da specie a specie. Nei procarioti
la quantità di DNA è molto inferiore a quella degli eucarioti, e viene
valutata in millesimi di pg.
|
Numero
dei cromosomi e quantità di DNA in pg |
|||
|
|
Genere o Specie |
Cromosomi |
DNA |
|
Procarioti |
|
|
|
|
|
Mycoplasma |
1 |
0,00084 |
|
|
Escherichia coli |
1 |
0,004 |
|
Eucarioti |
|
|
|
|
Protozoi |
Ameba proteus |
»
500 |
80 |
|
|
Plasmodium berghei |
¾ |
0,12 |
|
Fanerogame |
Crepis capillaris |
12 |
4,2 |
|
|
Allium cepa |
16 |
40 |
|
|
Lilium longiflorum |
24 |
100 |
|
Insetti |
Drosophila melanogaster |
8 |
0,18 |
|
|
Drosophila hydei |
12 |
0,2 |
|
|
Chironomus tentans |
8 |
0,5
|
|
|
Melanoplus differentialis |
24 |
16 |
|
Pesci |
Cyprinus |
104 |
3,2 |
|
|
Esox |
18 |
1,6 |
|
|
Amia |
46 |
2 |
|
Anfibi |
Rana esculenta |
26 |
14 |
|
|
Bufo bufo |
22 |
12 |
|
|
Triturus cristatus |
24 |
42 |
|
|
Necturus maculosus |
38 |
164 |
|
Rettili |
Testudo graeca |
52 |
10 |
|
|
Caiman crocodylus |
42 |
5,4 |
|
|
Lacerta campestris |
38 |
4,4 |
|
|
Boa costrictor |
36 |
6,6 |
|
Uccelli |
Gallus domesticus |
78 |
2,4 |
|
Mammiferi |
Homo sapiens |
46 |
7 |
|
|
Canis familiaris |
78 |
5,6 |
|
|
Bos taurus |
60 |
6,4 |